nnUNet的安裝
安裝python虛擬環境(作者不建議使用conda環境)
sudo apt-get install -y python3-venv
或
pip install virtualenv
建立虛擬環境資料夾
選擇要將Python程式設計環境放入哪個目錄,或者我們可以使用來建立一個新目錄mkdir
mkdir environments
進入資料夾
cd environments
建立虛擬環境
python3 -m venv project_env
project_env是虛擬環境的名字,建議命為nnunet, 方便記憶
或
virtualenv nnunet --python=python3.8指定版本
使用時需要啟用該環境
source nnunet/bin/activate
啟用要先切換到nnunet的上一級資料夾(也就是environment資料夾)
要離開環境,只需鍵入命令deactivate
安裝nnUNet
在虛擬環境中鍵入pip install nnunet
pip list檢視安裝了哪些包
設定環境變數
這一步對於nnUNet來講很重要,nnU-Net需要知道您打算將原始資料,預處理資料和訓練好的的模型儲存在何處
進入檔案bashrc檔案vim ~/.bashrc
按i鍵對檔案進行修改
將以下行放入檔案最後
export nnUNet_raw_data_base="/media/fabian/nnUNet_raw"
export nnUNet_preprocessed="/media/fabian/nnUNet_preprocessed"
export RESULTS_FOLDER="/media/fabian/nnUNet_trained_models"
/media/fabian/改為你自己的地址,下面要有這三個資料夾
Esc + :+ wq 退出並儲存修改之後的檔案
成功設定後,您可以通過鍵入echo $RESULTS_FOLDER etc來驗證路徑設定正確,並且應該列印出正確的資料夾
注意
安裝nnU-Net將向您的終端新增幾個新命令。這些命令用於執行整個nnU-Net管道。您可以從系統上的任何位置執行它們。
所有nnU-Net命令的字首都有 nnUNet_ 這樣易於識別。如訓練命令nnUNet_train
請注意,這些命令僅執行python指令碼。如果在虛擬環境中安裝了nnU-Net,則在執行命令時必須啟用該環境。
所有nnU-Net命令都有一個-h選項,該選項提供有關如何使用它們的資訊
私有資料預處理
nnU-Net的所有資料集都存放在nnUNet_raw/nnUNet_raw_data資料夾(上面安裝nnU-Net時指定了該資料夾的地址)
建立Task資料夾
在nnUNet_raw_data資料夾下建立Task066_LiverTumer資料夾(舉例說明,改成你自己的ID和name)
統一命名為(“Task”“ID”_“任務名稱”), “ID”是必須是三位數的整數,066而不是66
在每一個Task資料夾裡,又包含以下檔案
imagesTr: 訓練影像資料夾。
imagesTs(可選): 測試影像資料夾
labelTr: 訓練集標籤資料夾。
dataset.json : 包含資料集的後設資料的JSON檔案, 如任務名字,模態,標籤含義,訓練集包含的影像地址等
影像的處理
影像的統一命名格式case_identifier_XXXX.nii.gz, 標籤的統一命名格式case_identifier.nii.gz
例如mr_001_0000,mr_001_0001 => mr_001
可以寫一個python檔案將自己原本資料集裡的資料匯入並重新命名為標準格式(必須是從nii壓縮為nii.gz,不能是重新命名)
可以在資料夾用終端開啟,用gzip *可將資料夾內所有不是.gz的檔案壓縮為.gz
要注意每個影像所有的模態必須相同,不能少
json檔案
資料統一格式錄入資料夾後,生成json檔案,這裡可以參考我寫的(需要留言,比較懶)
開跑
資料驗證
驗證通過會自動進行下一步預處理,如果已經驗證過,可以去掉--之後的內容,直接開始預處理
66那裡填上自己的ID
nnUNet_plan_and_preprocess -t 66 --verify_dataset_integrity
訓練
nnUNet_train CONFIGURATION TRAINER_CLASS_NAME TASK_NAME_OR_ID FOLD --npz (additional options)
--npz不需要softmax輸出時不要加,會佔用大量空間,詳情見github說明
舉例:
nnUNet_train 2d nnUNetTrainerV2 66 4
66 處填寫任務ID
nnUNetTrainerV2 是要用到的使用的model trainer
4 是五折交叉驗證的第幾折(可選0,1,2,3,4)
2D U-Net:
For FOLD in [0, 1, 2, 3, 4], run:
nnUNet_train 2d nnUNetTrainerV2 TaskXXX_MYTASK FOLD --npz
3D full resolution U-Net
For FOLD in [0, 1, 2, 3, 4], run:
nnUNet_train 3d_fullres nnUNetTrainerV2 TaskXXX_MYTASK FOLD --npz
3D U-Net cascade
3D low resolution U-Net
For FOLD in [0, 1, 2, 3, 4], run:
nnUNet_train 3d_lowres nnUNetTrainerV2 TaskXXX_MYTASK FOLD --npz
3D full resolution U-Net
For FOLD in [0, 1, 2, 3, 4], run:
nnUNet_train 3d_cascade_fullres nnUNetTrainerV2CascadeFullRes TaskXXX_MYTASK FOLD --npz
注意:cascade的3D全解析度UNet需要事先完成低解析度UNet的五折
經過培訓的模型將被寫入RESULTS_FOLDER/nnUNet資料夾。每次訓練都會獲得自動生成的輸出資料夾名稱:
nnUNet_preprocessed/CONFIGURATION/TaskXXX_MYTASKNAME/TRAINER_CLASS_NAME__PLANS_FILE_NAME/FOLD
1000輪太多了,自定義epoch
修改這裡:
在nnunet虛擬環境裡找到nnunet資料夾
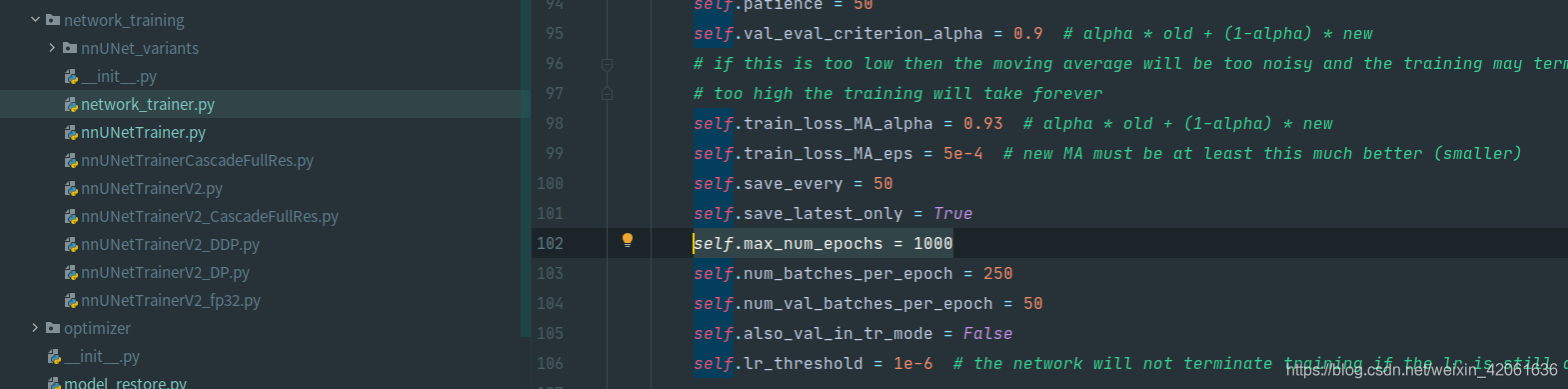
不起作用,修改這裡:
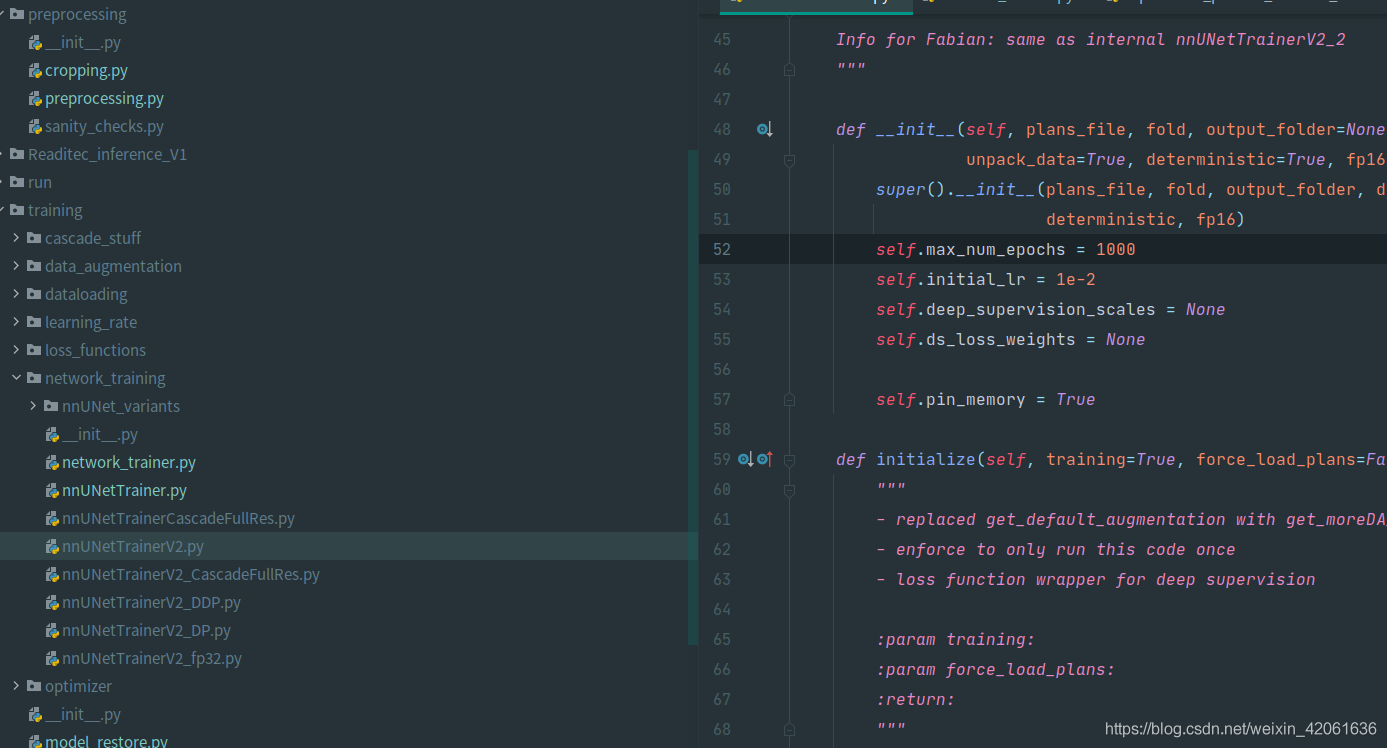
預測
nnUNet_predict -i INPUT_FOLDER -o OUTPUT_FOLDER -t TASK_NAME_OR_ID -m CONFIGURATION
INPUT_FOLDER: 測試資料地址
OUTPUT_FOLDER: 分割資料存放地址
CONFIGURATION: 使用的什麼架構,2d or 3d_fullres or 3d_cascade_fullres等
舉例:
nnUNet_predict -i /home/.../nnunet_file/nnUNet_raw/nnUNet_raw_data/Task066_LiverTumer/imagesTs -o /home/.../nnunet_file/output -t 66 -m 2d -f 4
參考:
不用寫程式碼神器!教你用4行命令輕鬆使用nnUNet訓練自己的醫學影像分割模型
nnUNet最舒服的訓練教程(讓我的奶奶也會用nnUNet(上))